Toujours dans le cadre de mes travaux de recherches, je devais comparer deux façons de calculer un NDVI cumulé annuel, 1) en intégrant toutes mes valeurs bi-mensuelles sur toute l'année, 2) en intégrant mes valeurs correspondant uniquement à la saison des pluies, c'est à dire environ de Mai à Octobre (contexte africain, au Niger).
Mes données d'entrée sont donc deux rasters calculés:
- raster1 = la manière 1 et comptant autant de bandes que d'années (ici 11)
- raster2 = la manière 2 et comptant autant de bandes que d'années (ici 11)
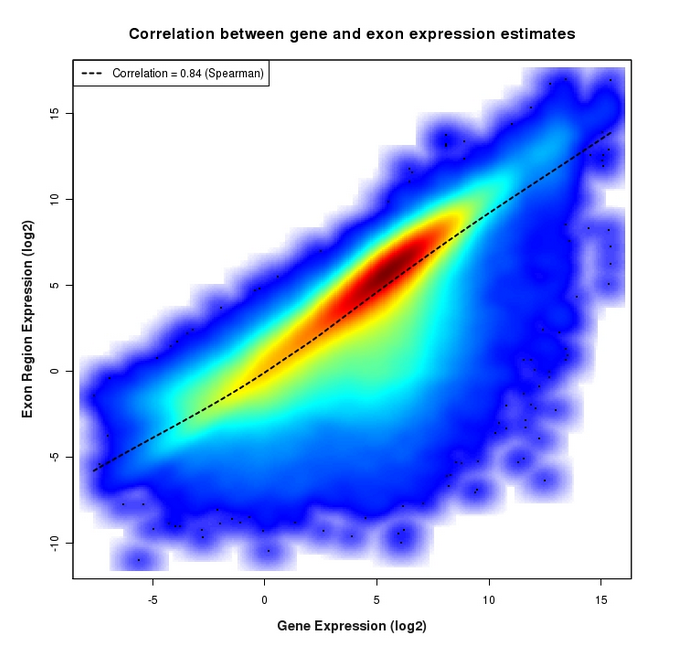
Le résultat que je souhaitais obtenir en sortie était quelque chose comme ceci :
mais avec un graphique par année (ou par bande).
J'ai donc décidé de faire cela sur …. R (^^).
Voici donc le code que j'ai mis en place. Dans mon cas j'avais besoin de le faire tourner sur plusieurs zones donc sur plusieurs images, il est donc fait pour tourner sur plusieurs paires de fichiers, mais en supprimer la première boucle vous pouver le faire tourner sur uniquement deux fichiers.
######### Comparaison de rasterS multibandes #######
# input : 2 Rasters multibandes format ENVI (dans ce cas si, mais fonctionne avec autres formats raster)
# output : graphique au format pdf de la "corrélation" de la bande 1…n d'un raster1 avec la bande 1…n d'un raster2
# Leroux Louise, CIRAD, UMR TETIS 2014
######## Initialisation #########
## Chargement de packages nécéssaires
library(raster)
library(sp)
library(Hmisc)
## Initialisation de l'espace de travail
setwd("D:/MyFolder")
##Importation des données raster##
# Pour le premier raster
liste.file1<-list.files(path=".", pattern="motif1")
liste.file1.hdr<-liste.file1[-grep(« .hdr », liste.file1, fixed=T)] #Récupération du nom sans l'extension
# Pour le second raster
liste.file2<-list.files(path=".", pattern="motif2")
liste.file2.hdr<-liste.file2[-grep(« .hdr », liste.file2, fixed=T)] #Récupération du nom sans l'extension
## Boucle sur les couples de fichiers ##
for (j in 1:length(liste.file1.hdr)){
name1<-liste.file1.hdr[j]
name2<-liste.file2.hdr[j]
nband<-nbands(raster(name1)) #variable stockant le nombre de bandes dans l'image
cell<-ncell(raster(name1)) #variable stockant le nombre de pixel d'une bande
mat1<-matrix(0, ncol=nband, nrow=cell)
mat2<-matrix(0, ncol=nband, nrow=cell)
# Stockage des bandes du raster 1 dans un matrice
for (i in 1:nband){
mat1[,i]<-getValues(raster(name1, band=i))
}
# Stockage des bandes du raster 2 dans un matrice
for (l in 1:nband){
mat2[,l]<-getValues(raster(name2, band=l))
}
## Création du graphique ##
out_name<-paste(out_name, ".pdf",sep="")
pdf(out_name, height=10,width=10)
ye<-seq(2000,2010,1) # Séquence pour nommer les graphiques, ici, 1 bande= 1 année
par(mfrow=c(4,3)) # Préparation de la fenêtre graphique : 4 lignes, 3 colonnes pour mettre un graphique par bande dans chaque case
# Boucle sur les bandes
for (k in 1:nband){
df <- data.frame(mat1[,i],mat2[,k])
x <- densCols(mat1[,k],mat2[,k], colramp=colorRampPalette(c("black", "white")))
# Création d'une palette pour représenter la concentration des individus
df$dens <- col2rgb(x)[1,] + 1L
cols <- colorRampPalette(c("#000099", "#00FEFF", "#45FE4F","#FCFF00", "#FF9400", "#FF3100"))(256)
df$col <- cols[df$dens]
plot(mat2[,k]~mat1[,k], data=df, pch=20, col=col, xlab=name_xlab, ylab=name_ylab, main=ye[k], cex.lab=0.75)
reg<-lm(mat2[,k]~mat1[,k])
abline(coef=coef(reg), lwd=1)
}
dev.off()
}
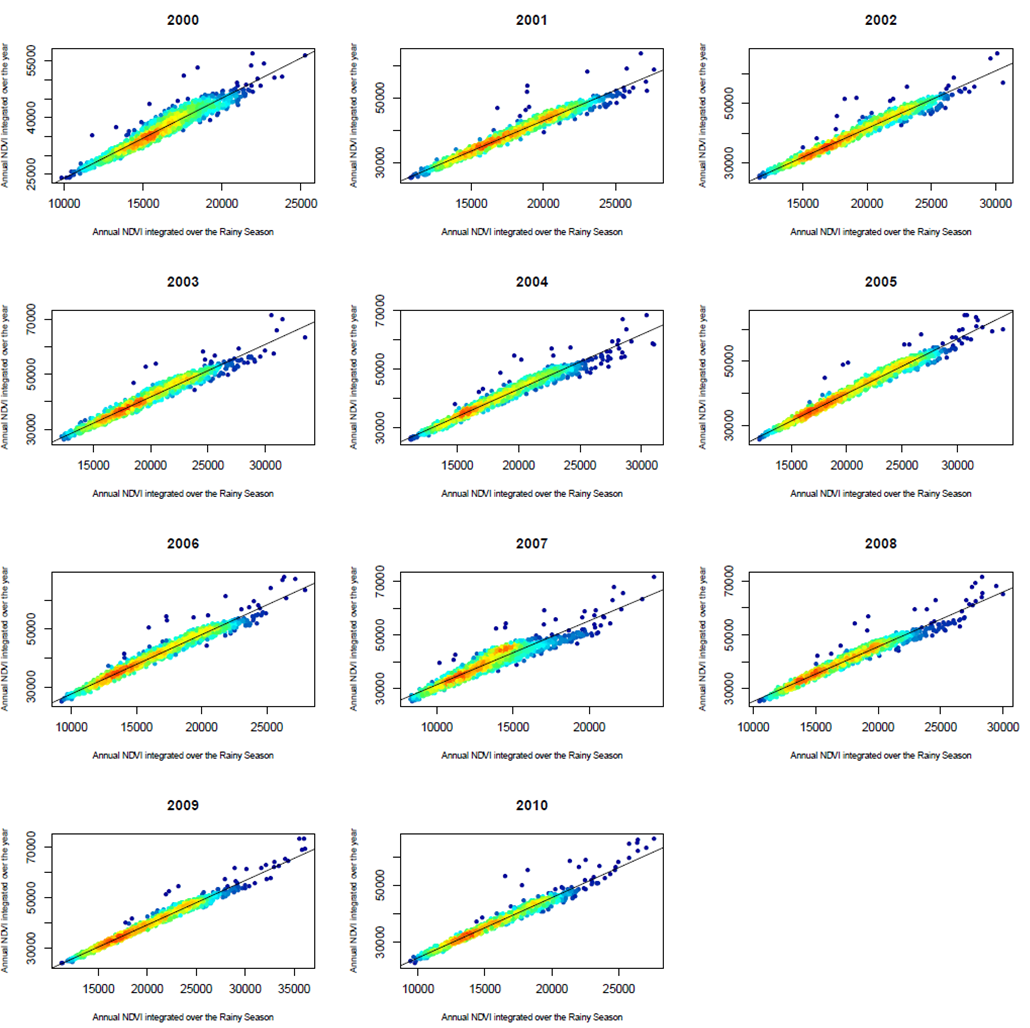
Et voici le résultat en sortie pour l'un de mes fichiers :